我正在尝试创建一个图表,显示点中给定组的测量值以及平均值和 95% CI。 R 不断告诉我它已删除值为 0 的行,因此其余点已关闭。
我使用的代码是:
data.summary <- data %>%
group_by(condition, conc) %>%
summarise(
sd = sd(score, na.rm = TRUE),
score = mean(score))
ggplot(data, aes(condition, score, color=concentration)) +
geom_jitter(aes(color=conc),position = position_jitter(0.2)) +
expand_limits(x = 0, y = 0) +
scale_color_manual("Conc",breaks = c("4", "5", "6", "7"),
values=c("sienna1", "dodgerblue1", "green1", "deeppink1"))+
new_scale("color")+
geom_pointrange(aes(ymin = score-sd, ymax = score+sd, color=conc),data = data.summary) +
expand_limits(x = 0, y = 0) +
scale_color_manual("Conc",breaks = c("4", "5", "6"),
values=c("sienna4", "dodgerblue4", "green4", "deeppink4")) +
ggtitle("Data")
数据(输出格式):
structure(list(hydra = c(1L, 2L, 3L, 4L, 5L, 6L, 7L, 8L, 9L,
1L, 2L, 3L, 4L, 5L, 6L, 7L, 8L, 9L, 1L, 2L, 3L, 4L, 5L, 6L, 7L,
8L, 9L, 1L, 2L, 3L, 4L, 5L, 6L, 7L, 8L, 9L, 1L, 2L, 3L, 4L, 5L,
6L, 7L, 8L, 9L, 1L, 2L, 3L, 4L, 5L, 6L, 7L, 8L, 9L, 1L, 2L, 3L,
4L, 5L, 6L, 7L, 8L, 9L, 1L, 2L, 3L, 4L, 5L, 6L, 7L, 8L, 9L),
time = c(72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L, 72L,
72L, 72L), condition = structure(c(1L, 1L, 1L, 1L, 1L, 1L,
1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L,
3L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L,
3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L, 4L, 4L, 4L,
4L, 4L, 4L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L,
4L, 4L, 4L, 4L, 4L, 4L), .Label = c("Control Bisection",
"Control Decapitation", "Treatment Bisection", "Treatment Decapitation"
), class = "factor"), conc = c(0L, 0L, 0L, 0L, 0L, 0L, 0L,
0L, 0L, 0L, 0L, 0L, 0L, 0L, 0L, 0L, 0L, 0L, 4L, 4L, 4L, 4L,
4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 5L,
5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L,
5L, 5L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L,
6L, 6L, 6L, 6L, 6L), score = c(9L, 10L, 9L, 7L, 10L, 9L,
9L, 9L, 10L, 9L, 9L, 10L, 6L, 9L, 10L, 10L, 10L, 9L, 2L,
0L, 3L, 0L, 0L, 5L, 0L, 0L, 0L, 7L, 5L, 4L, 3L, 3L, 3L, 4L,
5L, 3L, 0L, 0L, 0L, 0L, 3L, 0L, 2L, 0L, 2L, 0L, 4L, 2L, 3L,
0L, 7L, 5L, 3L, 4L, 0L, 5L, 3L, 4L, 2L, 0L, 4L, 0L, 4L, 4L,
4L, 3L, 5L, 2L, 3L, 4L, 5L, 4L)), class = "data.frame", row.names = c(NA,
-72L))
这就是数据应该是什么样子,与我得到的数据
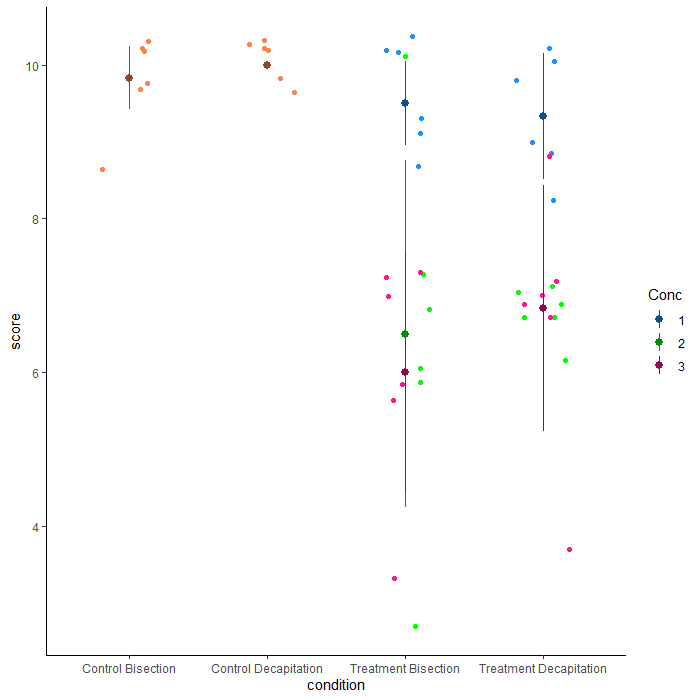

最佳答案
关于您的代码,有几件事没有按预期工作,但我认为主要问题是这一行:
scale_color_manual("Conc",breaks = c("4", "5", "6", "7"),
values=c("sienna1", "dodgerblue1", "green1", "deeppink1"))
您尚未指定浓度为零时该怎么办的值。这是可行的(我认为您实际上并不想要黑点,但您可以根据需要更改第一种颜色):
brkvec <- as.character(c(0, 4:7)
colvec <- c("black","sienna1", "dodgerblue1", "green1", "deeppink1")
scale_color_manual("Conc",breaks = brkvec, values = colvec)
关于r - geom_point 不接受 0 作为值,我们在Stack Overflow上找到一个类似的问题: https://stackoverflow.com/questions/62478322/