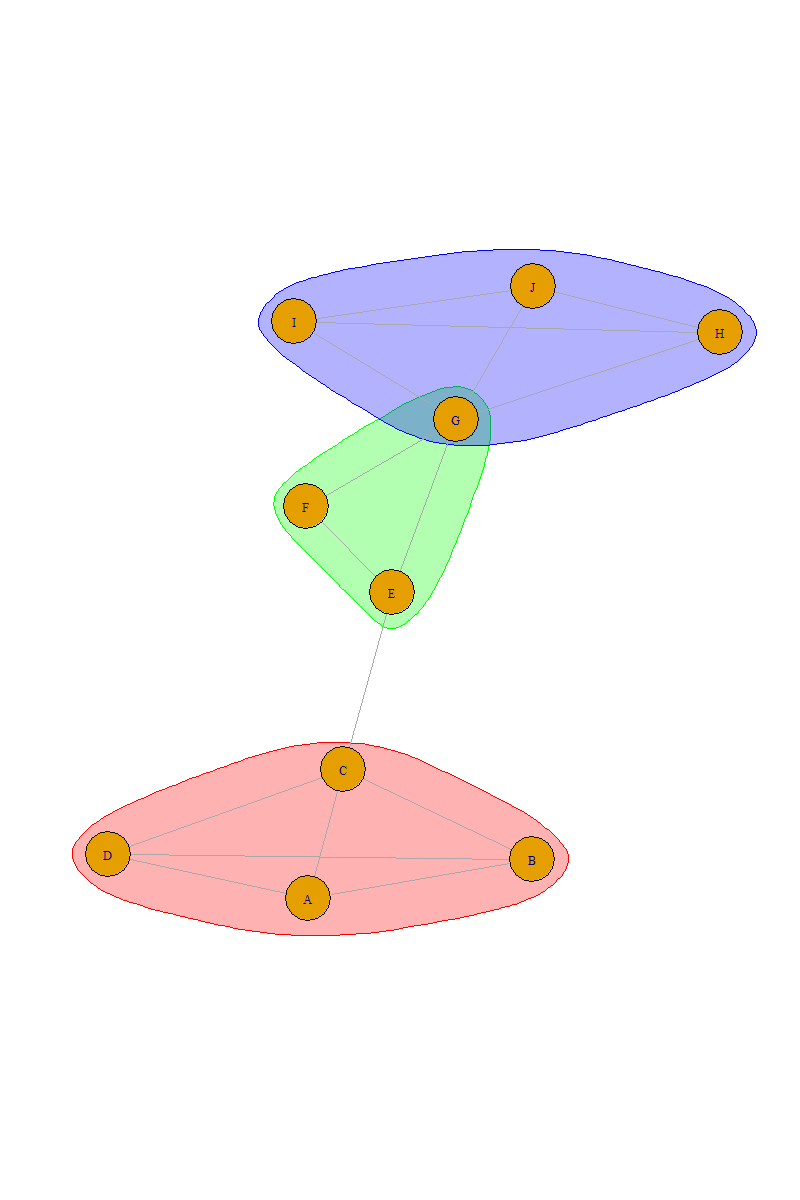
如果图表中存在重叠社区,如何可视化社区? 我可以使用 python(networkx、igraph、matplotlib 等)或 R 中的任何模块。
例如,节点、边以及每个社区中的节点的信息如下。请注意,节点 G 跨越两个社区。
list_nodes = ['A', 'B', 'C', 'D','E','F','G','H','I','J']
tuple_edges = [('A','B'),('A','C'),('A','D'),('B','C'),('B','D'), ('C','D'),('C','E'),
('E','F'),('E','G'),('F','G'),('G','H'),
('G','I'), ('G','J'),('H','I'),('H','J'),('I','J'),]
list_communities = [['A', 'B', 'C', 'D'],['E','F','G'],['G', 'H','I','J']]
我想要一个将社区可视化的图,如下所示。
在networkx中,可以对每个节点进行颜色编码,例如this post ,但当社区重叠时,此方法不适合。
在 igraph 中,可以使用包中包含的社区提取方法来可视化社区,如 this post 中所述。 。但是,就我而言,我想使用每个社区中包含的节点列表来定义社区。
最佳答案
下面是 R 中 igraph 的一个选项。
我认为您可能需要手动注释社区信息(请参阅下面的 grp),然后在绘图时使用它,例如,
g <- graph_from_data_frame(df, directed = FALSE)
grp <- lapply(
groups(cluster_edge_betweenness(g)),
function(x) {
c(
x,
names(which(colSums(distances(g, x) == 1) > 1))
)
}
)
plot(g, mark.groups = grp)
或使用 max_cliques 且尺寸最小为 3(感谢 @clp 的评论)
g <- graph_from_data_frame(df, directed = FALSE)
grp <- max_cliques(g, min = 3)
plot(g, mark.groups = grp)
数据
df <- data.frame(
from = c("A", "A", "A", "B", "B", "C", "C", "E", "E", "F", "G", "G", "G", "H", "H", "I"),
to = c("B", "C", "D", "C", "D", "D", "E", "F", "G", "G", "H", "I", "J", "I", "J", "J")
)
关于python - 通过任何 python 或 R 模块可视化图中重叠的社区,我们在Stack Overflow上找到一个类似的问题: https://stackoverflow.com/questions/74609794/