我正在尝试通过 Python 使用图论,以验证分子片段是否是较大分子的有效子结构。让我们看一个神经递质血清素的例子:
from pysmiles import read_smiles
import networkx as nx
import matplotlib.pyplot as plt
# Descriptor of serotonin, via SMILES notation
serotonin = "C1=CC2=C(C=C1O)C(=CN2)CCN"
# Uses pysmiles to convert the SMILES into a Graph object
serotonin_graph = read_smiles(serotonin)
# Function for plotting Graph objects of molecules
def plot_graph(graph):
# Small dictionary of how elements should be coloured
colors_dict = {"C": "grey", "N": "green", "O": "red"}
# Finds elements corresponding to nodes in the graph
elements = nx.get_node_attributes(graph, name = "element")
# Defines a list of colours for the nodes, accordingly
element_colors = [colors_dict[elements[e]] for e in elements]
# Displays the molecular graph
nx.draw(graph, node_color=element_colors, with_labels=True, labels=nx.get_node_attributes(graph, name = "element"))
plt.show()
# Runs the plotting function for serotonin
plot_graph(serotonin_graph)
# First three SMILES are valid fragments of serotonin, whereas the last is not
fragments = ['Oc:1:c:c:[c]:[c]:c1', 'NC[CH2]', 'CNCC', 'NC[CH2]:O:O:C']
# Converts fragments into Graph objects and displays them
for frag in fragments:
fragment_graph = read_smiles(frag)
plot_graph(fragment_graph)
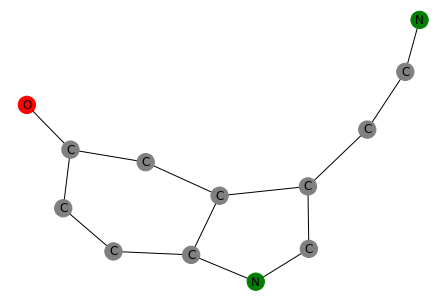
在本例中,我们正在处理血清素,上面的代码可能会将其绘制成分子图,如下所示(请注意,我们使用的符号自动忽略氢原子):
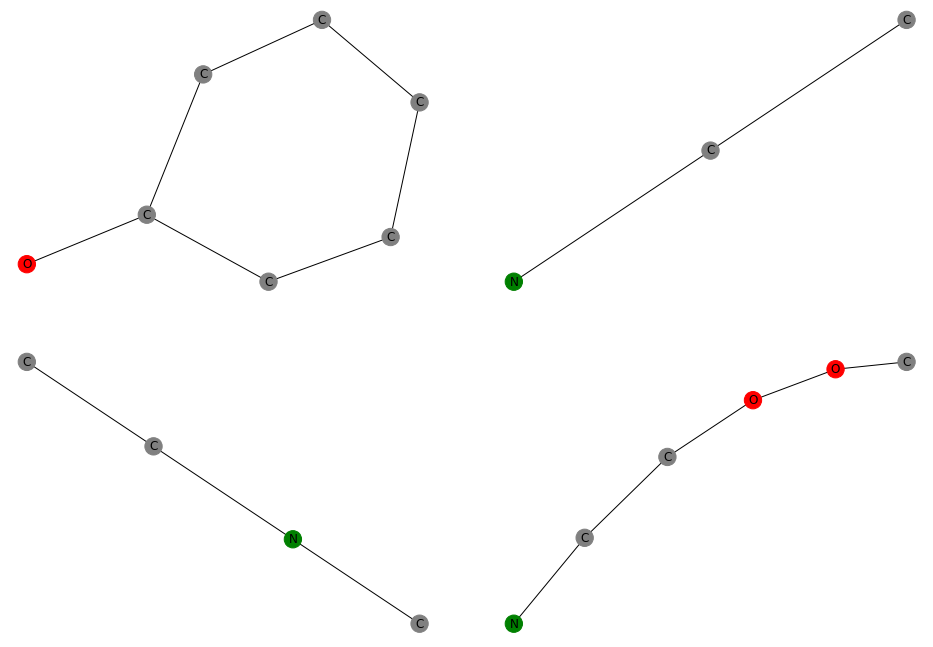
代码中考虑的 4 个分子片段也可以绘制成图表,如下所示:
通过肉眼可以轻松推断出,前三个是血清素的有效子图,而最后一个(右下)则不是 - 例如,最后一个片段包含两个氧原子,而血清素图显然不包含。
因此,我希望获得一个能够执行以下操作的函数:
>>> subgraph_checker(serotonin_graph, valid_substructure)
True
>>> subgraph_checker(serotonin_graph, invalid_substructure)
False
因此,除了最后一个右下角的片段图之外,该函数将为上面的所有片段图返回 True。
这里已经提出了关于使用 NetworkX 进行子图搜索的类似问题,但到目前为止还没有一个足够 - 例如有些解决方案特定于有向图,或推荐无效的解决方案。
这是我尝试过的一种解决方案,但它不起作用,使用 NetworkX 的函数来搜索同构子图:
GM = nx.algorithms.isomorphism.GraphMatcher(serotonin_graph, fragment_graph)
print(GM.subgraph_is_isomorphic())
当将上述应用于所有片段时,所有片段都返回 True,当然最后一个片段不应该出现这种情况。
我还尝试了替代解决方案,例如使用 RDKit 函数来直接搜索血清素内的子结构,但这些解决方案效果不佳,而且似乎是一个错误。
非常感谢您通过 NetworkX 使用图论解决此问题的任何帮助或见解!
最佳答案
您仅通过查看图的结构来检查同构。相反,在您的情况下,您还需要检查每个节点的内容。换句话说,您应该定义两个节点(无边)何时相等。我认为在你的情况下,这两个节点共享相同的“元素”就足够了。
for frag in fragments:
fragment_graph = read_smiles(frag)
gm = nx.isomorphism.GraphMatcher(
serotonin_graph,
fragment_graph,
node_match=lambda n1, n2: n1.get('element') == n2.get('element'),
)
print(gm.subgraph_is_isomorphic())
这是输出:
True
True
True
False
node_match 是一个返回 True 的函数,当且仅当在同构测试期间第一个图中的节点 n1 和第二个图中的节点 n2 应被视为相等。该函数将被调用如下:
node_match(G1.nodes[n1], G2.nodes[n2])。
也就是说,该函数将接收正在考虑的节点的节点属性字典。如果无,则在测试同构时不考虑任何属性。
更多信息可以找到here 。请注意,您还可以定义 edge_match 函数。
关于python - NetworkX 化学 : how to check if a smaller molecular graph A is a valid subgraph of a larger molecular graph B?,我们在Stack Overflow上找到一个类似的问题: https://stackoverflow.com/questions/73530124/