我正在尝试通过以下函数在数据集上通过素食包应用 NMDS:
library(vegan)
library(MASS)
library(MVA)
library(ggplot2)
all_d <- vegdist(landmetr, method= "hor", binary=FALSE, diag=TRUE,
upper=FALSE, na.rm = FALSE)
allmds3 <- isoMDS(all_d, k=3)
gr0 <- factor(france100_7c$LAND_CODE)
plot(allmds3$points[,2:3], col=gr0, bg="grey", cex=0.2,pch=20)
# regress environmental variables on configuration of site points
provaenv3 <- envfit(allmds3$points[,2:3], landmetr,999, na.rm = FALSE, france100_7c$LAND_CODE)
# add the environmental regressions to the area plot
plot(provaenv3,add=T, cex=0.5)
#plot0
ordiellipse (allmds3, gr0, kind = "sd", conf=0.95, label = T)
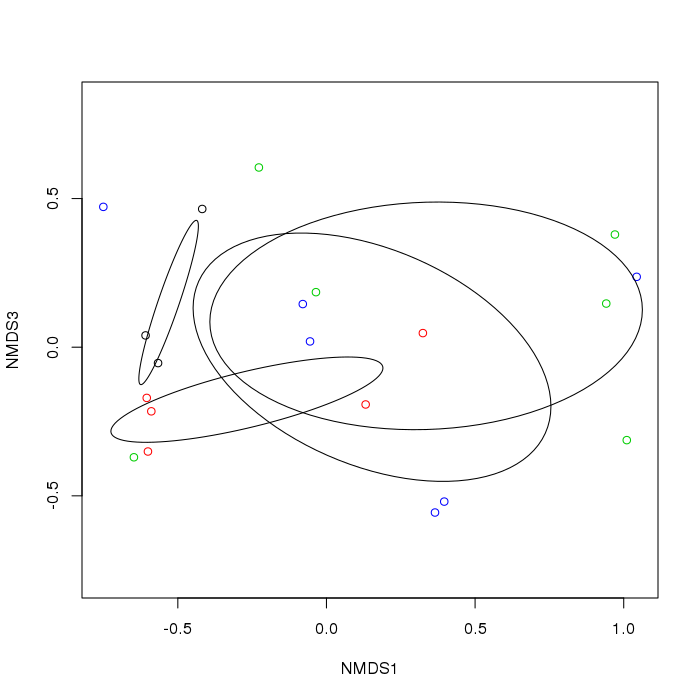
然而,尽管此双标图是在轴 2 和轴 3 上构建的,但 ordiellipse 函数写入椭圆以标识轴 1 和轴 2 双标图上的簇。 任何人都可以解释为什么会发生这种情况以及我如何在轴 2 和轴 3 双标图上绘制正确的椭圆?
最佳答案
在这种情况下,预期的用途是将 choices 也传递给 ordiellipse。如 ?ordiellipse
...: Parameters passed to graphical functions or to ‘scores’ to
select axes and scaling etc.
因此@EDi 的示例可以更简单地完成
library("vegan")
data(dune)
data(dune.env)
set.seed(1234)
NMDS <- metaMDS(dune, k = 3)
take <- c(1,3)
plot(NMDS, choices = take, display = "sites", type = "n")
points(NMDS, choices = take, display = "sites",
col = dune.env$Management)
with(dune.env, ordiellipse(NMDS, Management, choices = take))
生产
关于r - 在轴 2 和轴 3 上的 R 中的 NMDS 中使用 ordiellipse,我们在Stack Overflow上找到一个类似的问题: https://stackoverflow.com/questions/31426749/